SleepFM è un modello fondazionale multimodale per l’analisi del sonno che dimostra come una sola notte di polisomnografia possa diventare uno strumento predittivo avanzato per la salute futura. Dalla mortalità per tutte le cause alla demenza, dall’infarto miocardico all’ictus, SleepFM riesce a stimare il rischio di 130 condizioni cliniche con un livello di accuratezza che supera molti modelli specialistici tradizionali, aprendo uno scenario radicalmente nuovo per la medicina predittiva basata su segnali fisiologici.
L’elemento di rottura non è soltanto la quantità di malattie previste, ma il metodo: SleepFM non si affida a dati clinici strutturati o a storie sanitarie pregresse, bensì estrae informazione predittiva direttamente dalla struttura fisiologica e temporale del sonno, integrando attività cerebrale, segnali cardiaci, attività muscolare e respirazione. In questo modo il sonno smette di essere un indicatore secondario e diventa un vero sensore longitudinale di rischio biologico.
Architettura di SleepFM e approccio multimodale
SleepFM nasce come risposta a uno dei limiti storici dell’analisi del sonno basata su intelligenza artificiale: la frammentazione dei modelli, spesso addestrati su singole coorti, singole configurazioni di sensori o compiti molto specifici come lo staging del sonno o la diagnosi di apnea. Il modello adotta invece una architettura agnostica rispetto ai canali, progettata per funzionare su configurazioni polisomnografiche eterogenee, tipiche dei dataset clinici reali.
I segnali grezzi vengono acquisiti a 128 Hz e segmentati in finestre temporali di 5 secondi, che diventano i token di input del modello. Ogni modalità fisiologica è processata da un codificatore convoluzionale unidimensionale composto da sei strati, con un’espansione progressiva dei canali da 1 a 128, normalizzazione e attivazione ELU. Questo passaggio consente di catturare pattern locali complessi, come micro-arousal, variazioni di frequenza cardiaca o instabilità respiratoria.
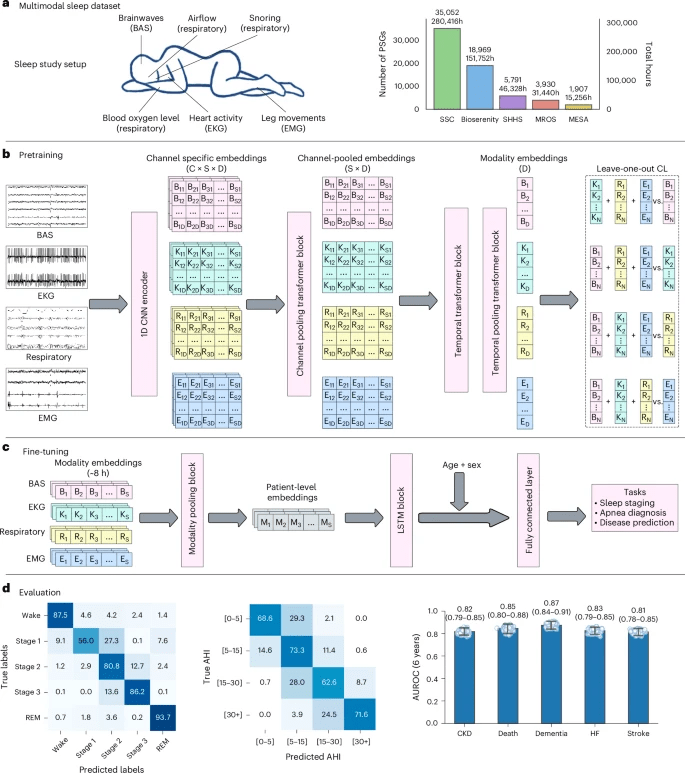
b , Preaddestramento contrastivo multimodale: i segnali grezzi di ciascuna modalità sono codificati da una CNN, gli embedding dei canali sono raggruppati all’interno della modalità e un trasformatore temporale con pooling temporale produce rappresentazioni a livello di sequenza per LOO-CL. C: canali, S: lunghezza della sequenza, D: dimensione dell’embedding.
c , Ottimizzazione tramite embedding congelati per attività a valle (stadiazione del sonno, rilevamento dell’apnea, previsione della malattia). Otto ore di embedding multimodali vengono aggregate in rappresentazioni a livello di paziente, concatenate con età e sesso e passate a un LSTM seguito da un livello completamente connesso.
d , Valutazione tra attività rappresentative e applicazioni cliniche. A sinistra e al centro: matrici di confusione per le categorie di stadiazione del sonno (SHHS) e AHI (SSC) mostrate come percentuali normalizzate per riga. A destra: performance di previsione della malattia nella coorte di Stanford (
n = 5.019 partecipanti). I box plot riassumono 1.000 ricampionamenti bootstrap a livello di paziente: punti deboli (disegni bootstrap individuali) e linea verticale con estremità (IC percentile bootstrap al 95%). Le etichette numeriche rappresentano le medie. Numero di campioni positivi per ciascuna malattia: CKD (354), decesso (224), demenza (221), scompenso cardiaco (283) e ictus (297).
La riduzione temporale avviene tramite pooling adattivo, che proietta i segnali in embedding da 128 dimensioni. Un elemento chiave è il pooling attention-based intra-modale, che rende l’architettura robusta a variazioni nel numero e nell’ordine dei canali tra diverse coorti cliniche. Successivamente, un trasformatore temporale a tre strati modella le dipendenze sequenziali su finestre di 5 minuti, integrando una codifica posizionale sinusoidale per preservare la struttura temporale del sonno.
Il risultato è un embedding multimodale compatto, informativo e trasferibile, che costituisce la base per il pre-addestramento auto-supervisionato e per i successivi compiti clinici.
Pre-addestramento contrastivo e apprendimento senza etichette
Il cuore concettuale di SleepFM è l’uso di un apprendimento contrastivo leave-one-out, una strategia che consente di sfruttare enormi quantità di dati non etichettati. Durante il pre-addestramento, il modello viene esposto a oltre 432.000 ore di registrazioni provenienti da circa 48.000 partecipanti, appartenenti a più coorti eterogenee.
Il principio è semplice ma potente: una modalità fisiologica viene esclusa e il modello è addestrato a predirne la rappresentazione latente a partire dalle altre. In questo modo SleepFM impara le relazioni strutturali tra segnali diversi, come il legame tra attività cerebrale e respirazione o tra variabilità cardiaca e stadi del sonno. La perdita contrastiva, basata su similarità coseno con temperatura scalabile, forza l’allineamento delle rappresentazioni multimodali nello spazio latente.
Questo approccio riduce drasticamente la dipendenza da annotazioni manuali, notoriamente costose e soggette a variabilità inter-esperto, e permette al modello di catturare pattern fisiologici fondamentali del sonno umano. Il pre-addestramento, completato in circa 15 ore su GPU A100, produce un modello con 4,44 milioni di parametri, sorprendentemente compatto rispetto ai foundation model di altri ambiti.
Prestazioni sui compiti standard di analisi del sonno
Un test cruciale per qualsiasi modello fondazionale è la sua capacità di competere con sistemi specializzati nei compiti classici. SleepFM dimostra prestazioni solide nello staging del sonno, distinguendo veglia, stadi N1, N2, N3 e REM su finestre di 5 secondi. Su coorti come Stanford, MESA, MrOS e SHHS, il modello raggiunge F1 medi tra 0,70 e 0,78, valori comparabili o superiori a sistemi consolidati come U-Sleep, YASA, GSSC e STAGES.
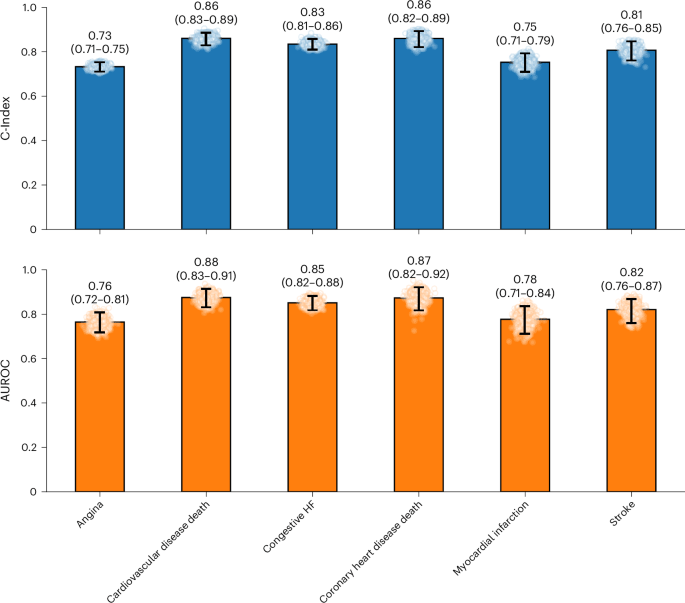
di P <0,01 dopo correzione di Bonferroni.
La robustezza emerge soprattutto negli stadi transizionali, dove anche l’annotazione umana presenta forte variabilità. La validazione esterna su dataset completamente esclusi dall’addestramento, come DCSM e HMC, conferma una buona capacità di generalizzazione, con F1 rispettivamente di 0,68 e 0,55, superando modelli baseline addestrati end-to-end.
SleepFM mostra inoltre buone prestazioni nella classificazione dell’apnea del sonno, raggiungendo un’accuratezza compresa tra 0,69 e 0,87 a seconda della severità e della coorte. Questi risultati indicano che il modello non sacrifica le capacità diagnostiche tradizionali a favore dei compiti predittivi, ma integra entrambe le dimensioni.
Predizione di 130 malattie da una sola notte di sonno
Il risultato più dirompente di SleepFM riguarda la predizione longitudinale delle malattie. Utilizzando embedding congelati e una fase di fine-tuning mirata, il modello viene addestrato a prevedere l’esordio futuro di 1.041 fenotipi clinici, mappati da codici EHR a phecodes, escludendo condizioni con prevalenza inferiore all’1,5%.
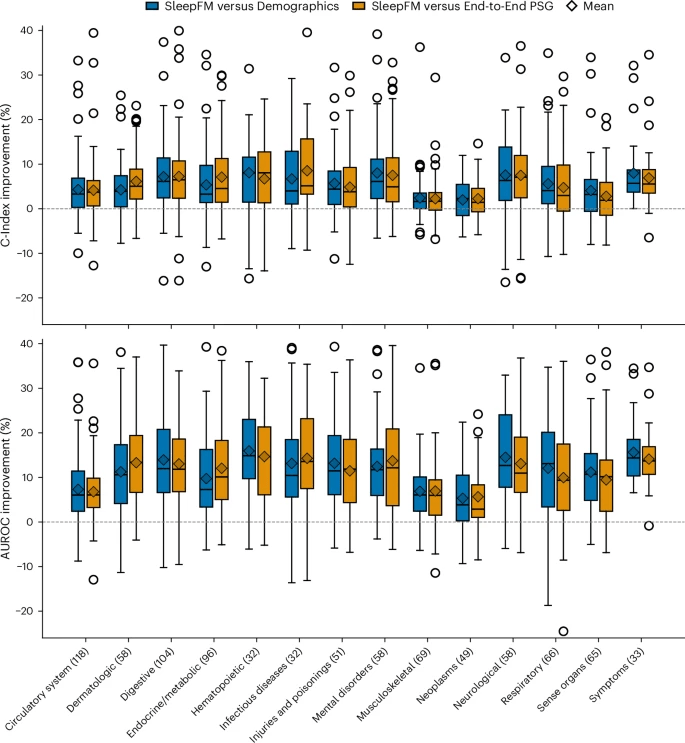
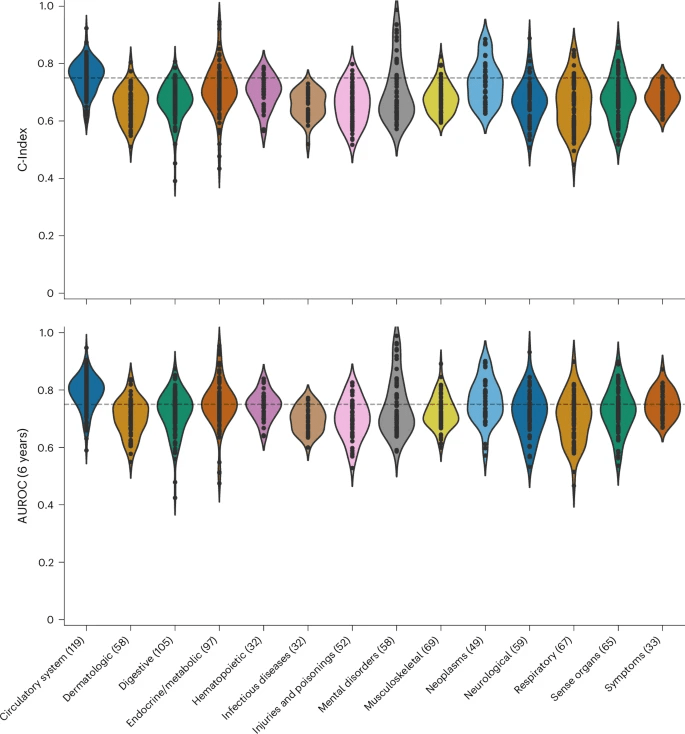
La funzione di perdita è basata su Cox proportional hazards multilabel, che consente di modellare il tempo all’evento per ciascuna condizione. I risultati mostrano che 130 malattie raggiungono un C-Index o AUROC ≥ 0,75, partendo da una singola registrazione notturna. Alcuni esempi emblematici includono una mortalità per tutte le cause a 0,84, demenza a 0,85, infarto miocardico a 0,81, insufficienza cardiaca a 0,80, malattia renale cronica a 0,79, ictus a 0,78 e fibrillazione atriale a 0,78.
Le prestazioni sono particolarmente elevate in ambiti neurologici e neurodegenerativi, con AUROC di 0,93 per il Parkinson e 0,84 per l’impairment cognitivo lieve, suggerendo che il sonno contenga segnali precoci di declino cerebrale ben prima della diagnosi clinica. Anche in oncologia emergono risultati rilevanti, con predizioni robuste per tumore alla prostata, tumore al seno e melanoma.
Generalizzazione temporale e trasferibilità clinica
Uno dei rischi principali dei modelli clinici basati su dati storici è la degradazione delle prestazioni nel tempo. SleepFM è stato testato su una coorte Stanford post-2020, completamente esclusa dall’addestramento, mantenendo C-Index significativi per mortalità, insufficienza cardiaca e demenza. Questo indica una resilienza ai cambiamenti nelle pratiche cliniche e nelle popolazioni di riferimento.
Ancora più rilevante è il test di transfer learning su nuove coorti, come lo Sleep Heart Health Study, escluso dal pre-addestramento. Con un fine-tuning minimo su circa 3.300 partecipanti e test su 2.000 soggetti, SleepFM predice condizioni cardiovascolari critiche come ictus, insufficienza cardiaca congestizia e mortalità cardiovascolare con AUROC fino a 0,88, simulando uno scenario di deployment reale con supervisione limitata.
Confronto con baseline e modelli end-to-end
SleepFM supera in modo sistematico i baseline demografici, basati su età, sesso, BMI ed etnia, con guadagni di AUROC tra il 5% e il 17%, particolarmente marcati per disturbi neurologici, ematopoietici e cardiovascolari. Anche rispetto a modelli end-to-end addestrati direttamente sui segnali grezzi senza pre-addestramento, SleepFM mostra un vantaggio netto, soprattutto in condizioni complesse come demenza senile, aterosclerosi e diabete con complicanze circolatorie.
Un aspetto cruciale è la scalabilità del fine-tuning: anche utilizzando solo il 10% dei dati, SleepFM supera baseline addestrati su dataset cinque volte più grandi, evidenziando l’efficienza degli embedding pre-addestrati.
Implicazioni cliniche e prospettive future
SleepFM ridefinisce il ruolo del sonno nella medicina digitale, dimostrando che le dinamiche notturne non sono solo un riflesso dello stato di salute attuale, ma un indicatore predittivo di traiettorie patologiche future. Questo apre la strada a sistemi di screening non invasivi, basati su una singola notte di registrazione, potenzialmente integrabili con EHR, dati omici e imaging. Restano tuttavia alcune sfide, tra cui il bias di selezione delle coorti cliniche, la necessità di maggiore interpretabilità caso-specifica e il rischio di degradazione su popolazioni completamente diverse. Le analisi stratificate per stadi del sonno e modalità fisiologiche rappresentano un primo passo verso una spiegabilità più fine, ma ulteriori sviluppi saranno necessari per un’adozione clinica su larga scala.
Domande frequenti su SleepFM
SleepFM può essere usato come strumento diagnostico clinico?
SleepFM non è progettato come strumento diagnostico diretto, ma come modello predittivo di rischio. Le sue stime indicano probabilità future di malattia e devono essere interpretate come supporto decisionale per il clinico, non come diagnosi automatica.
Perché il sonno è così informativo per la predizione delle malattie?
Il sonno integra segnali neurologici, cardiovascolari, respiratori e metabolici in modo continuo. Alterazioni sottili nella sua struttura riflettono disfunzioni sistemiche precoci, spesso invisibili negli esami clinici standard.
SleepFM funziona anche con configurazioni di sensori diverse?
Sì, l’architettura è progettata per essere agnostica rispetto al numero e all’ordine dei canali, rendendola adatta a dataset polisomnografici eterogenei e a contesti clinici reali.
Quali sono i principali limiti attuali di SleepFM?
I limiti principali riguardano la rappresentatività delle coorti, la complessità interpretativa delle predizioni e la necessità di validazioni prospettiche su larga scala prima dell’uso routinario in clinica.
Iscriviti alla Newsletter
Non perdere le analisi settimanali: Entra nella Matrice Digitale.
Matrice Digitale partecipa al Programma Affiliazione Amazon EU. In qualità di Affiliato Amazon, ricevo un guadagno dagli acquisti idonei. Questo non influenza i prezzi per te.