L’Arc Institute compie un passo rivoluzionario nella biologia sintetica progettando batteriofagi generati interamente mediante intelligenza artificiale, aprendo la strada a terapie antivirali di nuova generazione e a una comprensione più profonda dei processi evolutivi. Sotto la guida di Samuel H. King e del team di Brian L. Hie, i ricercatori hanno sviluppato modelli linguistici genomici denominati Evo 1 ed Evo 2, capaci di generare sequenze virali realistiche basate sul fago litico Φ X174. L’esperimento culmina nella creazione di 16 fagi vitali su 302 candidati sintetizzati, alcuni dei quali mostrano mutazioni mai osservate prima e fitness superiore rispetto al fago naturale di riferimento.
Una nuova era per la biologia sintetica
Il progetto segna la prima dimostrazione di design generativo di genomi virali completi: un risultato reso possibile grazie a modelli addestrati su milioni di genomi di batteriofagi e successivamente raffinati con dataset specifici della famiglia Microviridae. A differenza degli approcci di ingegneria genetica tradizionale, basati su modifiche razionali o su evoluzione diretta, i modelli Evo imparano le regole implicite della biologia virale — architetture geniche, vincoli di tropismo e meccanismi regolatori — per produrre nuove combinazioni plausibili di geni.
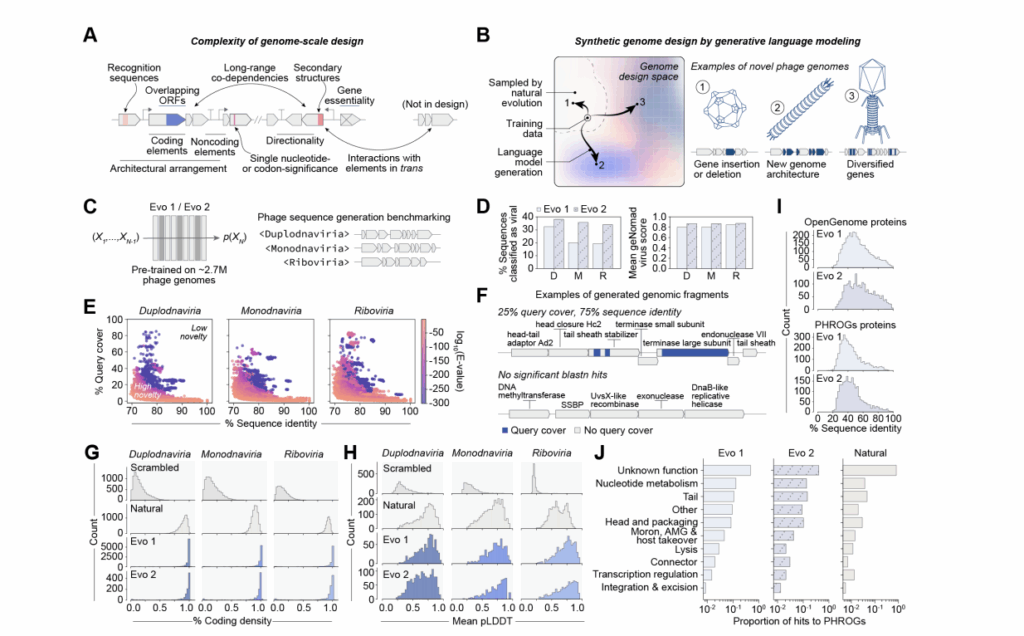
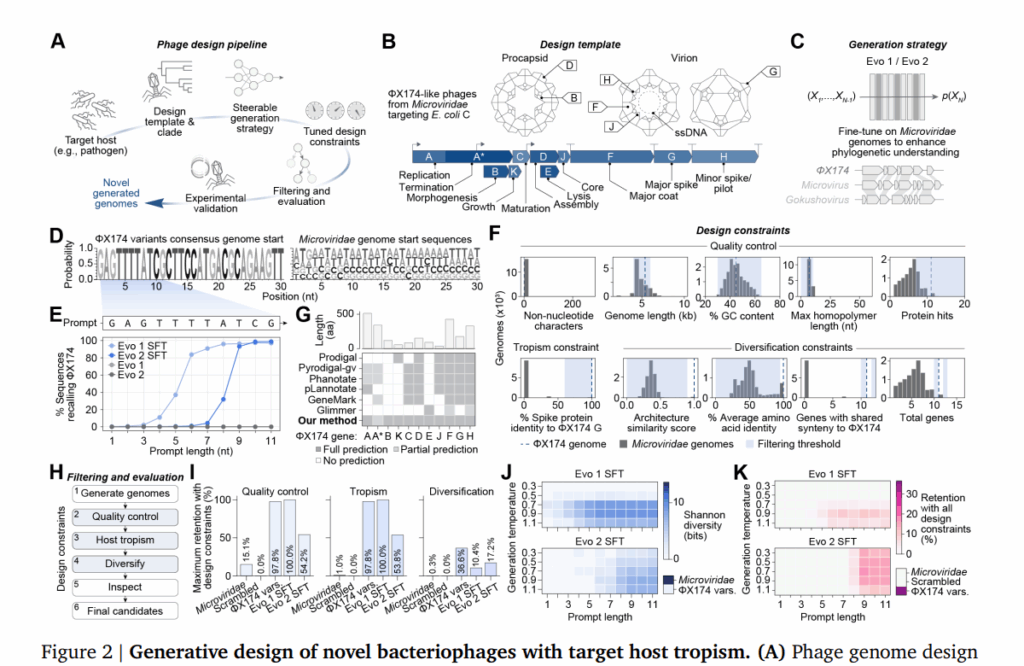
L’obiettivo iniziale era creare fagi vitali basati sul piccolo e storicamente importante Φ X174, un virus a DNA singolo di circa 5.4 kb che infetta Escherichia coli e rappresenta uno dei sistemi modello più studiati in biologia molecolare. Usando un addestramento senza supervisione, seguito da fine-tuning su sequenze affini, i ricercatori hanno guidato l’intelligenza artificiale verso la generazione di genomi funzionali, bilanciando vincoli di qualità (lunghezza e contenuto GC), tropismo (identità proteica con Φ X174) e diversità evolutiva.
Metodi di modellazione generativa
I modelli Evo 1 ed Evo 2 sono stati preaddestrati su oltre due milioni di genomi virali, quindi raffinati su 15.000 sequenze Microviridae ad alta identità. Attraverso prompt tassonomici e nucleotidici, i modelli autoregressivi hanno generato genomi tra 4 e 6 kb, controllando la coerenza funzionale e la variabilità.
Le sequenze sono state filtrate con tre livelli di vincoli:
- Qualità, per evitare omopolimeri e garantire coerenza genomica;
- Tropismo, mantenendo somiglianza strutturale con Φ X174 per assicurare infezione di E. coli;
- Diversificazione, limitando l’identità media per ottenere fagi nuovi ma compatibili.
Dopo la generazione, i genomi selezionati sono stati sintetizzati tramite Twist Biosciences e assemblati con Gibson e Golden Gate cloning. La validazione biologica su E. coli C ha confermato la vitalità di 16 varianti, ciascuna con proprietà uniche di replicazione, lisi e adattamento evolutivo.
Risultati genomici e strutturali
Le sequenze create mostrano alta densità codificante e architetture coerenti con i fagi naturali, ma introducono novità genetiche sostanziali. Alcuni genomi presentano fino a 392 mutazioni inedite, tra cui inserzioni e sostituzioni nei geni del capsid e delle proteine spike. In particolare, il fago Evo-Φ 2147 si distingue come nuova specie virale, con un’identità inferiore al 95% rispetto ai fagi conosciuti.
La microscopia cryo-EM a risoluzione di 2,8–2,9 Å rivela configurazioni capsidiche e proteine di impacchettamento evolutivamente distanti, confermando che l’intelligenza artificiale è in grado di generare strutture biologiche nuove ma funzionali. Le analisi condotte con AlphaFold 3 ed ESMFold mostrano stabilità comparabile a quella dei fagi naturali, con punteggi di confidenza elevati.
Fitness superiore e resistenza batterica
Nei test di competizione diretta, diversi fagi generati — tra cui Evo-Φ 69 ed Evo-Φ 2483 — hanno superato Φ X174 in velocità di replicazione e capacità litica, con differenze di fitness fino a 65 volte superiori. La cinetica di lisi evidenzia un declino più rapido della densità ottica (OD600) e un tempo di clearing di appena 135 minuti. Quando impiegati in cocktail terapeutici, i fagi generativi hanno eliminato rapidamente ceppi di E. coli resistenti al fago naturale, adattandosi grazie a ricombinazioni e mutazioni compensatorie. Questi risultati aprono la strada a terapie fagogiche evolutivamente resilienti, capaci di contrastare l’emergere di resistenze batteriche.
Sicurezza e prospettive
Il team sottolinea la biosicurezza dell’esperimento: i modelli sono stati addestrati esclusivamente su virus batterici, evitando dati relativi a virus eucariotici. Tutte le procedure si sono svolte in condizioni BSL-1, con tropismo controllato per ridurre qualsiasi rischio biologico. Questa ricerca rappresenta una prova di concetto per la progettazione generativa di sistemi viventi. L’approccio potrà estendersi alla progettazione di genomi più complessi, con applicazioni che spaziano dalla terapia antimicrobica alla sintesi di circuiti biologici su scala genomica.
Prospettive
La creazione di fagi funzionali generati da modelli linguistici introduce una nuova disciplina — la genomica generativa — che unisce intelligenza artificiale e biologia sintetica. L’approccio non solo amplia la comprensione dei limiti evolutivi, ma fornisce un framework replicabile per progettare nuovi organismi. Gli autori vedono nel futuro la possibilità di addestrare modelli su dataset più ampi e diversificati, migliorando il condizionamento per tropismi specifici e integrando moduli evolutivi adattivi. Questo lavoro pone le basi per un design computazionale di organismi viventi, dove l’intelligenza artificiale non si limita più a prevedere la vita, ma inizia a crearla.
Iscriviti alla Newsletter
Non perdere le analisi settimanali: Entra nella Matrice Digitale.
Matrice Digitale partecipa al Programma Affiliazione Amazon EU. In qualità di Affiliato Amazon, ricevo un guadagno dagli acquisti idonei. Questo non influenza i prezzi per te.